Alle Welt spricht von dem neuen Genome‐Editing‐Werkzeug. Doch woher kommt die »Genschere« ursprünglich, in welchen Lebewesen ist sie zu finden, und welche Funktionen erfüllt sie in der Natur?
Auf einen Blick
- 1
Das CRISPR/Cas‐System revolutioniert seit einigen Jahren die Gentechnik. Es ist einfacher zu handhaben, billiger und flexibler als frühere Methoden des Genome Editing.
- 2
Die öffentliche Debatte dreht sich meist um die biotechnologischen Anwendungen des Systems. Nicht minder interessant sind aber die damit verbundenen grundlegenden biologischen Fragen.
- 3
Forscher haben bereits eine Menge über CRISPR/Cas herausgefunden, doch es gibt noch viele Geheimnisse zu lüften.
Francisco Mojica kann sich noch gut an den Tag im Jahr 1992 erinnern, an dem er erstmals einen Eindruck vom CRISPR/Cas‐System bekam – jenem Immunmechanismus von Bakterien und Archaeen, der 20 Jahre später eine Revolution in der Biotechnologie lostreten sollte. Mojica war damit beschäftigt, Genomsequenzen der Salz liebenden Mikrobe Haloferax mediterranei auszuwerten, die zu den Archaeen gehört. Dabei fielen ihm 14 ungewöhnliche DNA‐Sequenzen auf, die jeweils 30 Nukleotide lang waren. Ob vorwärts‑ oder rückwärtsgelesen – sie sahen immer mehr oder weniger gleich aus. Und sie wiederholten sich in einem Abstand von etwa 35 Nukleotiden. Wenig später fand Mojica noch mehr von ihnen. Der Forscher war fasziniert und stellte die mysteriösen Sequenzen ins Zentrum seiner Forschung, die er an der spanischen Universidad de Alicante betrieb.
Eine populäre Entscheidung war das nicht. Das Labor des Mikrobiologen musste jahrelang mit kargen Finanzmitteln zurechtkommen. Auf Tagungen trat er an bedeutende Forscher heran und fragte sie, was sie von den kurzen, sich wiederholenden Sequenzen hielten. »Vergeuden Sie nicht zu viel Zeit damit«, warnten sie ihn, »es gibt so viele repetitive DNA‐Sequenzen in so vielen Organismen – wer kann schon sagen, was es damit auf sich hat?«
Mittlerweile können wir eine Menge darüber sagen. Was Mojica seinerzeit in den Bann zog, bezeichnen wir heute als gehäuft auftretende, in regelmäßigen Abständen angeordnete, kurze palindromische Wiederholungen (clustered, regularly interspaced palindromic repeats, kurz CRISPR; als »palindromisch« bezeichnet man DNA‐Abschnitte, wenn ihre beiden Stränge gegenläufig die gleiche Sequenz aufweisen). Sie gehören zum so genannten CRISPR/Cas‐System, das Mikroorganismen dabei hilft, eindringende Viren zu zerstören. Die meisten Biologen und Mediziner wissen inzwischen zu schätzen, wie gut sich mit dem System – insbesondere seiner Version CRISPR/Cas9 – Gensequenzen verändern lassen.
Doch Mojica und andere Mikrobiologen rätseln noch immer über grundlegende Fragen: Wie ist das System im Lauf der Evolution entstanden, und wie hat es die Entwicklung der Mikroorganismen geprägt? Warum nutzen manche Mikroben es, andere dagegen nicht? Und erfüllt es möglicherweise noch weitere biologische Funktionen als die, die man schon kennt?
»CRISPR/Cas hat viel Aufmerksamkeit in den Medien bekommen, hauptsächlich wegen seiner Verwendung als Genome‐Editing‐Werkzeug – und das aus gutem Grund, schließlich liegt hierin eine außerordentlich große gesellschaftliche Bedeutung«, sagt Jennifer Doudna, Molekularbiologin an der University of California in Berkeley. Sie gehörte zu den ersten Wissenschaftlerinnen, die das biotechnologische Potenzial von CRISPR/Cas erkannten. »Doch kaum weniger spannend sind die vielen grundsätzlichen Fragen zur einschlägigen Biologie, die immer noch einer Antwort harren.«
Woher kommt CRISPR/Cas?
Die evolutionären Vorteile dieses molekularen Systems liegen auf der Hand. Bakterien und Archaeen, die beide zu den Prokaryoten zählen, müssen sich ständig gegen Angriffe von Eindringlingen wehren. Viren beispielsweise übertreffen Prokaryoten zahlenmäßig mindestens um das Zehnfache – und vernichten schätzungsweise alle zwei Tage die Hälfte sämtlicher Mikroben auf dem Planeten (Letztere vermehren sich natürlich immer wieder, so dass sie nicht verschwinden). Hinzu kommen parasitische DNA‐Plasmide, die per interzellulärem Austausch von einem Mikroorganismus zum anderen wechseln, ihrem Wirt wichtige Ressourcen stehlen und ihn zur Selbstzerstörung zwingen, falls er versucht, sie wieder loszuwerden.
Im Zuge der Evolution haben die Prokaryoten ein ganzes Arsenal von Waffen entwickelt, um solchen Bedrohungen zu begegnen. Mit bestimmten Proteinen, den Restriktionsenzymen, zerschneiden sie etwa fremde DNA. Aber das ist häufig ein stumpfes Schwert, denn jedes Restriktionsenzym erkennt lediglich eine spezifische Sequenz, und ein Mikroorganismus kann sich nur dann vor einem Schädling schützen, wenn er das dazu passende Enzym besitzt.
Verglichen damit erweist sich CRISPR/Cas als wesentlich flexibleres Verteidigungssystem. Es passt sich an Eindringlinge an und erinnert sich später wieder an sie – und zwar auf ähnliche Weise, wie menschliche Antikörper nach einer Infektion für lang anhaltende Immunität sorgen. Mojica und andere erkannten, dass die DNA‐Sequenzen zwischen den palindromischen Wiederholungen, die »Spacer«, manchmal mit Abschnitten im Erbgut von Viren übereinstimmen. Es stellte sich heraus: Kommt eine Mikrobe mit Viren oder Plasmiden in Kontakt, fügen einige ihrer Proteine kurze Stücke aus deren DNA‐Sequenz als Spacer ins Zellgenom ein. Diese Proteine stehen mit CRISPR in Zusammenhang und werden deshalb als Cas‐Proteine bezeichnet (von »CRISPR‐associated«). Der zelluläre Ableseapparat der Mikrobe setzt die eingefügten Spacer in RNA‐Moleküle um, die ihrerseits dazu dienen, andere Cas‐Proteine zu Eindringlingen passender Sequenz hinzuleiten. Die Cas‐Proteine zerschneiden das fremde Erbgut dann.
Wie kommt es, dass Bakterien und Archaeen einen solch raffinierten Abwehrmechanismus besitzen? Das ist nicht abschließend beantwortet, aber vermutlich stammt das System von Transposons ab: »springenden Genen«, die ihre Position im Genom verändern können. Der Evolutionsbiologe Eugene Koonin von den US‐amerikanischen National Institutes of Health in Bethesda (Maryland) und sein Team haben eine Klasse von Transposons entdeckt, die für das Protein Cas1 kodieren, welches am Einbau der Spacer ins mikrobielle Genom mitwirkt. Diese »Casposons« stellen vielleicht den Ursprung des CRISPR/Cas‐Systems dar.
Wie funktioniert CRISPR/Cas?
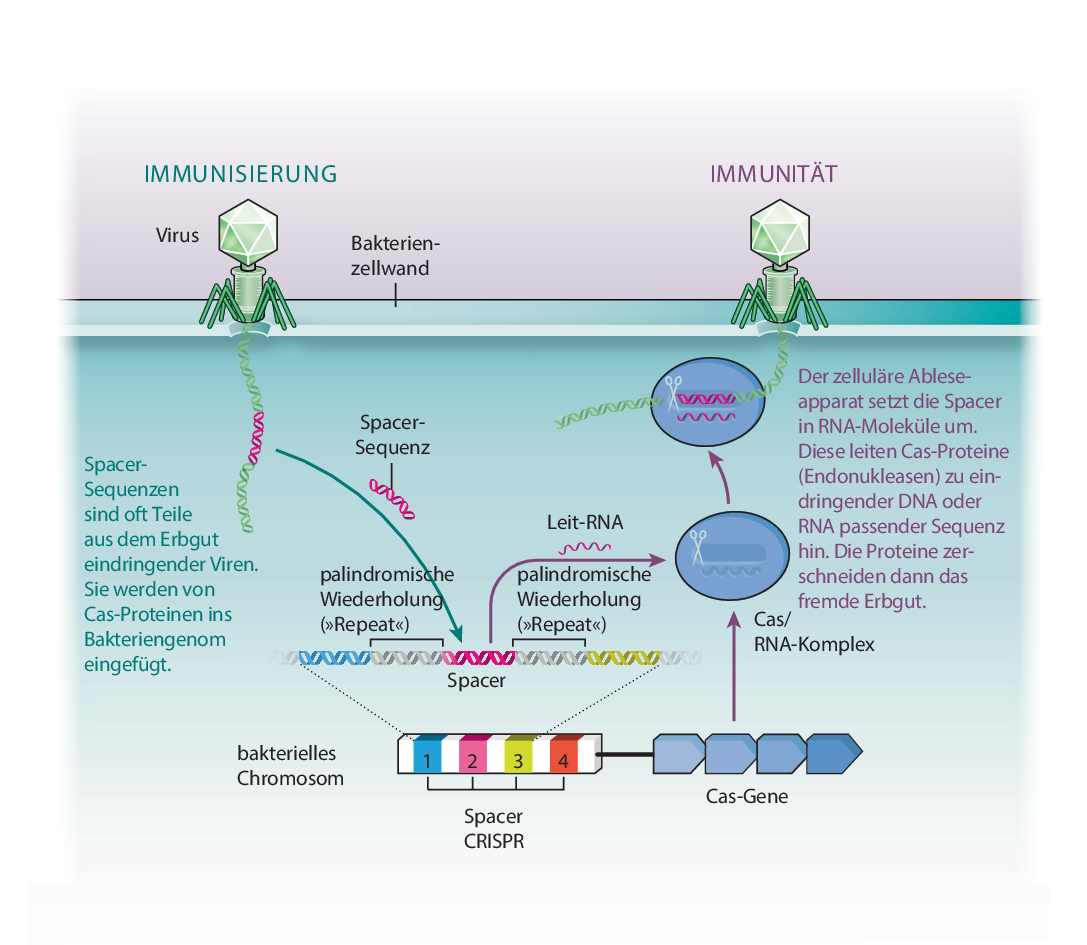
Dauerhafter Schutz. Etwa 90 Prozent der bekannten Archaeen und gut ein Drittel der Bakterien besitzen irgendeine Form von CRISPR/Cas‐System. Es besteht oft aus gehäuft auftretenden, kurzen DNA‐Wiederholungssequenzen mit Spacer‐Sequenzen dazwischen sowie aus einigen Genen, die für CRISPR‐assoziierte Proteine (Cas‐Proteine) kodieren. (Nik Spencer/Nature; Ledford, H.: Crispr’s Mysteries. In: Nature 541, S. 280–282, 2017)
Der Mikrobiologe Rodolphe Barrangou von der North Carolina State University in Raleigh vermutet, dass solche Fehler durchaus vorkommen, aber nicht die Gesamtpopulation der Wirtszellen auslöschen. Ein paar tödliche Versehen fallen wohl nicht ins Gewicht, wenn dafür andere Zellen einen Virenangriff überstehen und anschließend immunisiert sind.
Wenn Viren eine Bakterienpopulation befallen, erwirbt oft nur eine von zehn Millionen Zellen einen Spacer, mit dem sie sich anschließend erfolgreich verteidigen kann. Das erschwert es, die Details des Vorgangs aufzuklären. Dabei wäre es nützlich, mehr darüber zu erfahren und etwa dafür zu sorgen, dass bestimmte Spacer häufiger eingefügt werden. Forschungsarbeiten haben gezeigt: Zellen mit funktionierendem CRISPR/Cas‐System können DNA‐ und RNA‐Sequenzen speichern, mit denen sie in Kontakt kommen. Hier eröffnet sich die Möglichkeit, zelluläre Automaten zu konstruieren, die Umwelteinflüsse aufzeichnen.
Forscher wüssten auch gern, warum CRISPR‐assoziierte Sequenzen häufig wieder aus dem Zellgenom verschwinden. Die meisten Mikroorganismen, die über ein CRISPR/Cas‐System verfügen, besitzen nur wenige Dutzend Spacer – manche sogar lediglich einen einzigen. Der krasse Gegensatz dazu ist Sulfolobus tokodaii, ein Vertreter der Archaeen, dessen fünf CRISPR/Cas‐Systeme rund ein Prozent seines gesamten Genoms belegen und 458 Spacer enthalten.
Der Selektionsdruck, alte Spacer beizubehalten, dürfte eher schwach sein. Wenn ein Virus mutiert, so dass die Sequenz seines Erbguts nicht mehr mit derjenigen des entsprechenden Spacers übereinstimmt, können sich die potenziellen Wirtszellen nicht mehr vor dem Virus schützen. Der Spacer wird dann überflüssig und für die Zelle zum genetischen Ballast.
Welche Funktionen könnte CRISPR/Cas noch haben?
Bei vielen Spacern ist rätselhaft, woher sie stammen. Nicht einmal drei Prozent der bisher untersuchten stimmen mit bekannten DNA‐Sequenzen in Datenbanken überein. Das könnte unserem begrenzten Wissen geschuldet sein: Bislang haben Forscher vor allem solche Viren analysiert, die Menschen, Nutztiere oder Nutzpflanzen infizieren – kaum aber solche, die Bakterien oder gar Archaeen befallen. Möglich erscheint auch, dass manche Spacer die »Geister« längst nicht mehr existierender oder bis zur Unkenntlichkeit mutierter infektiöser Partikel sind.
Besonders fasziniert die Forscher ein dritter Aspekt. Wie sich herausgestellt hat, gibt es bakterielle CRISPR/Cas‐Systeme, die nicht nur genetische Eindringlinge abwehren, sondern darüber hinaus die DNA‐Reparatur, die Genexpression und die Bildung von Biofilmen kontrollieren. Sie können zudem die Infektiosität von Bakterien beeinflussen: Legionella pneumophila, der Erreger der Legionärskrankheit, benötigt das Protein Cas2, um Amöben zu befallen, die seine natürlichen Wirte sind. Es stellt sich die Frage, in welchem Umfang die Funktionen von CRISPR/Cas‐Systemen über die reine Schädlingsabwehr hinausgehen. Hier gibt es interessante Parallelen zur RNA‐Interferenz, einem Vorgang, der es Lebewesen mit Zellkern erlaubt, ihre Gene abzuschalten. Von ihm dachten Forscher anfangs ebenfalls, er sei vorrangig ein Abwehrmechanismus; erst später fiel auf, dass er an der Steuerung der Genexpression mitwirkt.
Laut dem Mikrobiologen Stan Brouns von der Technischen Universität Delft (Niederlande) können CRISPR/Cas‐Strukturen wohl alle möglichen DNA‐Sequenzen als Spacer aufnehmen. Manchmal entstünden dabei neue, nützliche Funktionen, manchmal gingen die Zellen daran zu Grunde.
Warum nutzen nur manche Mikroben CRISPR/Cas?
Schätzungen zufolge besitzen mehr als 90 Prozent der Archaeen ein CRISPR/Cas‐basiertes Abwehrsystem, während es bei den bisher sequenzierten Bakterien lediglich rund 30 Prozent sind. Bei Eukaryoten, also Lebewesen mit Zellkern, hat man CRISPR/Cas bislang überhaupt noch nicht gefunden, nicht einmal bei Einzellern.
Nanoarchaeum equitans ist ein Organismus aus der Domäne der Archaeen, der in nahezu kochendem Wasser auf anderen Archaeen parasitiert. Er hat sich vieler Gene entledigt, die mit dem Energiehaushalt und der allgemeinen Zellorganisation zu tun haben. Doch selbst sein winziges Genom, dessen Sequenz gerade einmal 490.000 Nukleotide umfasst, hat ein CRISPR/Cas‐System mit ungefähr 30 Spacern. Anscheinend ist es sogar für diesen Zwergorganismus unverzichtbar – warum es dann in Eukaryoten komplett fehlt, bleibt zu klären.
Möglicherweise fördern extreme Umweltbedingungen den Unterhalt eines CRISPR/Cas‐Systems, weil sie kaum andere Verteidigungsmethoden zulassen. Bakterien entziehen sich Eindringlingen oft, indem sie Mutationen in jenen Sequenzen erwerben, die für Proteine an ihrer Oberfläche kodieren. Extremophilen Archaeen könnte dieser Weg versperrt sein, da ihre Oberflächenproteine weniger Freiheiten haben, sich zu verändern, ohne dass die Zellen dabei ihre Lebensfähigkeit einbüßen.
Es dürften noch weitere Faktoren eine Rolle spielen. Mycoplasma gallisepticum beispielsweise, ein Krankheitserreger bei Vögeln, verlor seine CRISPR/Cas‐Ausstattung, als er von Hühnern auf Wildfinken übersprang. Warum das System in dem alten Wirt anscheinend nützlich war, in dem neuen aber plötzlich nicht mehr, ist ein Rätsel.
Mathematische Modelle und vorläufige experimentelle Befunde deuten darauf hin, dass CRISPR/Cas vor allem dann Vorteile bringt, wenn ein Organismus sich nur mit wenigen Virustypen auseinandersetzen muss. Denn das System kann bloß eine begrenzte Zahl viraler Sequenzen speichern, bevor die damit einhergehende zusätzliche DNA‐Menge für den Organismus zur Belastung wird. Übersteigt die Vielfalt der Viren in der Umwelt die Zahl der möglichen Spacer bei Weitem, nutzt CRISPR/Cas vermutlich nur noch wenig.
Wie viele CRISPR/Cas‐Varianten gibt es?
Öffentliche Debatten drehen sich meist um das System CRISPR/Cas9 – zu Recht, denn es ist vergleichsweise einfach zu handhaben, kostengünstig und vielseitig. Mikroorganismen jedoch haben keine Vorlieben. Sie nehmen neue Systeme von anderen Mikroben auf, vermischen sie mit ihren eigenen und entledigen sich dabei alter Elemente.
Forscher unterscheiden zwischen sechs CRISPR/Cas‐Typen, die sich in 19 Untertypen gliedern. Nur bei einigen davon ist geklärt, wie sie funktionieren. CRISPR/Cas9 etwa ist ein System des Typs II, das die Spacer‐Sequenzen in RNA‐Moleküle umsetzt, die dann ihrerseits ein Enzym (eine Endonuklease) zu eindringender Fremd‐DNA hinleiten, so dass es diese zerschneidet. Systeme des Typs VI dagegen funktionieren mit Enzymen, die RNA statt DNA zerlegen. Und solche des Typs IV enthalten zwar einige Gene, die mit CRISPR/Cas in Zusammenhang stehen, ihnen fehlen aber die palindromischen Wiederholungen und der Apparat zum Einbau der Spacer.
Typ‐III‐Systeme treten in der Natur am häufigsten auf und sind am wenigsten verstanden. Die bisherigen Befunde deuten darauf hin, dass sie nicht direkt auf eindringende DNA oder RNA reagieren, sondern auf den Prozess des Umschreibens von DNA in RNA, die so genannte Transkription. Falls das stimmt, würde es neue Möglichkeiten des Genome Editing eröffnen, wie Doudna betont. Möglicherweise harren noch weitere Systeme ihrer Entdeckung, insbesondere da Forscher nicht mehr nur in Mikroben nach ihnen suchen, welche in Kulturen gezüchtet wurden, sondern auch in DNA‐Proben aus der Umwelt (»environmental DNA«).
Mojica hält es für reizvoller, die biologischen Grundlagen der CRISPR‐Systeme zu erforschen, als ihre biotechnologischen Anwendungen weiterzuentwickeln. Viele seiner Kollegen wundern sich darüber, zumal für Letzteres wesentlich mehr Mittel bereitstehen. Doch Mojica bleibt dabei: »Ich weiß, dass es ein großartiges Biotechnologiewerkzeug mit großem medizinischem Potenzial ist – und dennoch interessiert mich mehr, wie es funktioniert.
Der Artikel erschien ursprünglich in Nature 541, S. 280–282.
Quellen
Nuñez, J. K. et al.: Integrase‐Mediated Spacer Acquisition during CRISPR‐Cas Adaptive Immunity. In: Nature 519, S. 193–198, 2015
Shipman, S. L. et al.: Molecular Recordings by Directed CRISPR Spacer Acquisition. In: Science 353, aaf1175, 2016
Westra, E. R. et al.: Parasite Exposure Drives Selective Evolution of Constitutive versus Inducible Defense. In: Current Biology 25, S. 1043–1049, 2015