Im letzten Kapitel hast du bereits Einsichten in Lernstrategien bekommen und mehrere unserer Beispiele nachvollzogen. Jetzt wollen wir uns zusammen weitere Details ansehen und dein Verständnis vertiefen. Was sind die wichtigen Eigenschaften der Daten? Was hängt womit zusammen? Auch etwas statistisches Wissen kommt hier hinzu.
Der ausführbare Code für dieses Kapitel inklusive der erzeugten Grafiken ist unter der URL https://github.com/DJCordhose/buch-machine-learning-notebooks (https://bit.ly/ml-kug) als Notebook kap5.ipynb erreichbar.
Am Anfang eines Projekts stehst du manchmal vor einem Haufen Daten mit sehr vielen Attributen pro Datensatz. Alle diese Attribute sind potenzielle Features, allerdings weißt du noch nicht, ob alle wirklich nützlich sind. Da man mit Sklearn relativ einfach mit multidimensionalen Daten arbeiten kann, könnte man beschließen, einfach alle Attribute als Feature-Input für einen Machine-Learning-Algorithmus zu benutzen.
Es gibt allerdings drei gute Gründe, warum eine Auswahl bzw. Reduzierung der Features häufig sinnvoll ist:
Punkt 1 wird oft auch als Fluch der hohen Dimensionen bezeichnet. Zur Illustration generieren wir mithilfe des Zufallsgenerators von NumPy drei Features: x1, x2 und x3. Sie werden dabei unabhängig voneinander so generiert, dass sie gleichförmig im Bereich zwischen 0 und 10 liegen:
import numpy as np
n = 100; vmin = 0; vmax = 10
x1 = np.random.uniform(vmin, vmax, n)
x2 = np.random.uniform(vmin, vmax, n)
x3 = np.random.uniform(vmin, vmax, n)
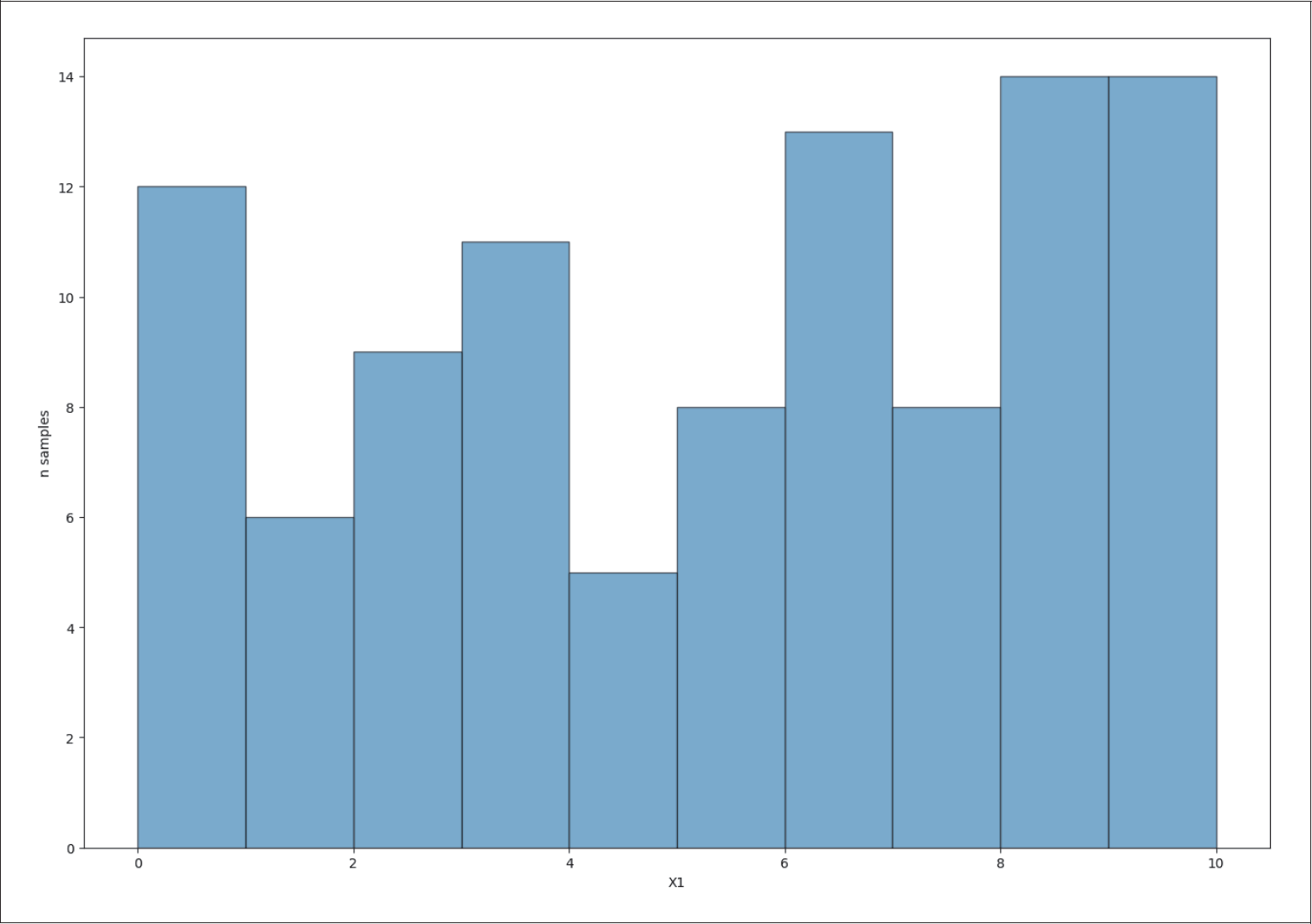
In Abbildung 5-1 ist die eindimensionale Verteilung von x1 als Histogramm dargestellt. Wir haben nun Bins, die jeweils einen Wertebereich zusammenfassen. Du kannst erkennen, dass ein Bin der Länge 1 im Durchschnitt etwa 10 Beispiele enthält, wenn wir 100 Beispiele generieren.
Abbildung 5-1: Verteilung eines Features x1
Nun erhöhen wir schrittweise die Dimension und sehen, wie sich das auf die Verteilung auswirkt. Die zweidimensionale Verteilung von x1 und x2 in Abbildung 5-2 zeigt schon eine deutliche »Verdünnung« der Beispiele pro Quadrat mit der Kantenlänge 1. Statistisch erwarten wir im Durchschnitt ein Beispiel pro Box.
Bei der dreidimensionalen Verteilung von x1, x2, x3 in Abbildung 5-3 erwarten wir nur noch 0,1 Beispiele pro Kubus mit der Kantenlänge 1. Das heißt, je mehr Features du benutzt, desto weniger Daten pro Einheitsbox im Feature-Raum hast du im Durchschnitt zur Verfügung.
Abbildung 5-2: Zweidimensionale Verteilung der Features x1 und x2
Abbildung 5-3: Dreidimensionale Verteilung der Features x1, x2, x3
Die statistische Signifikanz der Ergebnisse aller Machine-Learning-Algorithmen hängt sowohl von der Anzahl der Daten, die du analysierst, als auch von der Anzahl der Features ab, die du wählst. Eine unzureichende Anzahl von Daten führt häufig zu Overfitting. Benutzt du zu viele Features, steht durchschnittlich pro Raumeinheit eine zu kleine Anzahl an Stichproben zur Verfügung, sodass signifikante Aussagen nicht mehr möglich sind.
Den Zusammenhang zwischen der Anzahl der Trainingsbeispiele, der Komplexität eines Modells und Overfitting kann man am besten am Beispiel von nicht-linearer Regression zeigen. Dazu generieren wir eine kubische Verteilung (Polynom 3. Grades) mit 20 Datenpunkten (siehe Abbildung 5-4).
Abbildung 5-4: 20 Datenpunkte aus einer kubischen Funktion
Wenn du den Grad des Polynoms bereits vorher weißt, kannst du oft schon mit 20 Datenpunkten ein gutes Modell ohne Overfitting erhalten, wie du in Abbildung 5-5 sehen kannst.
Abbildung 5-5: Regression mit einem Polynom 3. Grades
In der Regel kennen wir die Komplexität unseres Problems aber nicht. Um Underfitting zu vermeiden, wird daher häufig eher ein zu komplexes Modell gewählt. In unserem Beispiel wäre eine lineare Regression (eine Gerade ist ein Polynom 1. Grades) ein zu simples Modell. Das erkennst du auch am Ergebnis der linearen Regression in Abbildung 5-6.
Abbildung 5-6: Underfitting mit linearer Regression
Ein zu komplexes Modell führt hingegen zu Overfitting. Im Extremfall wird sogar jeder Datenpunkt perfekt vorhergesagt, wenn wir z.B. ein Polynom 20. Grades zur Regression benutzen, wie in Abbildung 5-7 dargestellt.
Abbildung 5-7: Overfitting bei einer Regression mit einem Polynom 20. Grades
Abbildung 5-8: Regression mit einem Polynom 3. Grades mit vielen Daten
Wenn wir nun statt 20 200 Datenpunkte generieren, erhalten wir bei Regression mit einem Polynom 3. Grades wiederum eine gutes Modell, wie man in Abbildung 5-8 sehen kann.
Bei der Regression mit einem Polynom 20. Grades mit wesentlich mehr Daten als vorher sehen wir in Abbildung 5-9, dass das Overfitting nicht mehr ganz so schlimm ist wie vorher. Das Ergebnis des Modells ist sogar ganz passabel.
Abbildung 5-9: Regression mit einem Polynom 20. Grades mit vielen Daten
Für viele Probleme gilt in der Regel, dass man mit mehr Daten, weniger Features und geringerer Komplexität des Modells die Wahrscheinlichkeit des Overfittings reduziert.
Im vorigen Kapitel haben wir dir Regularisierungsmethoden für verschiedene Algorithmen gezeigt, mit denen man die Komplexität der Modelle kontrollieren kann. Die Anzahl der Daten ist oft begrenzt. Die Reduzierung der Features ist daher häufig eine gute Option zur Optimierung deiner Ergebnisse.
In diesem Kapitel werden wir dir einige Techniken zeigen, mit denen man die Anzahl der Features reduzieren kann, ohne dabei wichtige Informationen zu verlieren, bzw. mit denen wir den Informationsverlust minimieren können.
Im letzten Abschnitt haben wir darüber diskutiert, warum es wichtig ist, nur eine begrenzte Anzahl von Features zu benutzen. In den Fällen, bei denen es sinnvoll ist, die Anzahl der Features zu reduzieren, stellen sich also diese Fragen:
Die perfekte Feature-Kombination wird man bei vielen komplexeren Problemen nur schwer finden können, aber wir versuchen, dieser so nahe wie möglich zu kommen. Im Folgenden möchten wir dir anhand des Irisbeispiels zeigen, wie man systematisch an diese Fragen herangeht. Wir werden dir einige Methoden zeigen, mit deren Hilfe du abschätzen kannst, welche Features du benutzen und welche du weglassen kannst oder solltest.
Oft ergibt es Sinn, Features erst dann zu reduzieren, wenn sehr viele zur Auswahl stehen. Die obere Grenze hängt meist ab von deinem speziellen Problem, dem verwendeten Algorithmus, den vorhandenen Ressourcen etc. Es kann also schon bei zehn Features beginnen oder erst bei mehreren Hundert.
Der besseren Anschaulichkeit wegen werden wir keinen Datensatz mit vielen Features als Beispiel nehmen, sondern unseren Irisdatensatz mit vier Features. Wir wissen schon, dass alle vier Features informativ sind, d. h., jedes trägt dazu bei, eine bessere Klassifizierung zu erhalten. Zu Demonstrationszwecken werden wir für die Beispiele zusätzliche Features generieren, die entweder nicht informativ oder aus einem der anderen Features abgeleitet (also redundant) sind.
Am Anfang eines Projekts mit neuen Daten hilft es oft, verschiedene Diagramme zu erstellen. Dadurch erhältst du ein besseres Verständnis von den Daten, d.h. ein besseres Bild über die Zusammenhänge zwischen den verschiedenen potenziellen Features und dem Target. Bei den sogenannten univariaten Diagrammen werden Histogramme der einzelnen Features erstellt.
In unserem Irisdatensatz sind die Längen und Breiten der beiden Blütenblätterarten (setosa, versicolor, virginica) von 150 Irispflanzen aufgelistet: sepal length, sepal width, petal length, petal width. Die entsprechenden vier Histogramme sind in Abbildung 5-10 dargestellt.
Abbildung 5-10: Univariate Verteilungen des Irisdatensatzes
Wie du siehst, kann man die Art Iris setosa von den anderen beiden Irisarten sehr leicht mithilfe nur eines Features unterscheiden: entweder über die Petal-Länge oder Petal-Breite. Im folgenden Code wenden wir den linearen SVC für jeweils ein Feature an:
from sklearn.datasets import load_iris
import numpy as np
from sklearn.svm import SVC
svc = SVC(kernel='linear')
iris = load_iris()
for fid in range(len(iris.feature_names)):
X = iris.data[:, fid, np.newaxis]
y = iris.target
y[y==2] = 1 # map 2 → 1
clf = svc.fit(X, y)
print(iris.feature_names[fid], "(univariate score):", clf.score(X, y))
Dabei iterieren wir über die vier Feature-IDs und selektieren mit
X = iris.data[:, fid, np.newaxis]
jeweils nur ein Feature. Mit
y[y==2] = 1
fassen wir die Arten versicolor und virginica in einer Klasse zusammen und können damit über
clf = svc.fit(X, y)
die Art setosa von den Arten versicolor und virginica unterscheiden. Die Ergebnisse sehen so aus, wie wir es erwartet haben:
sepal length (cm) (univariate score): 0.893333333333
sepal width (cm) (univariate score): 0.826666666667
petal length (cm) (univariate score): 1.0
petal width (cm) (univariate score): 1.0
Mit petal length oder petal width können wir setosa von den beiden anderen Arten perfekt trennen, aber nicht mit sepal length oder sepal width.
Die Arten versicolor und virginica können hingegen sogar mit allen vier Features nicht gut getrennt werden. Das siehst du mithilfe dieses Codes, in dem wir die Arten versicolor und virginica explizit vorher herausfiltern:
iris = load_iris() # reload data
X = iris.data
y = iris.target
ids = y >= 1 # select only versicolor and virginica
print(clf.score(X, y))
Der Score ist nur 0,66.
Eine einfache Methode, um die Beziehung zwischen zwei Features oder einem Feature und dem Target zu verstehen, ist die Berechnung des Pearson-Korrelationskoeffizienten r, den wir schon kurz bei der Bestimmung von Ausreißern in Kapitel 3, Datenimport und -vorbereitung, gesehen haben. Er ist folgendermaßen definiert:
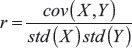
… wobei cov(X,Y) für die Kovarianz zwischen den Features X und Y steht und std(X) für die Standardabweichung von X (die genaue Definition kannst du auch hier nachschlagen: https://de.wikipedia.org/wiki/Korrelationskoeffizient).
Ein r-Wert von 1 bedeutet, dass die beiden Variablen linear 100% korreliert sind, ein Wert von –1, dass sie 100% linear antikorreliert sind, und ein Wert von 0, dass keine lineare Korrelation besteht.
Beispiele für einige Verteilungen mit den zugehörigen Korrelationskoeffizienten sind in Abbildung 5-11 dargestellt.
Abbildung 5-11: Pearson-Korrelationskoeffizient für verschiedene Verteilungen in der x-y-Ebene
(Abbildung aus https://en.wikipedia.org/wiki/Pearson_correlation_coefficient)
Starke Korrelation zwischen zwei Features ist ein Indikator dafür, dass die beiden Features möglicherweise ähnliche Informationen tragen.
In Abbildung 5-12 sind die sechs bivariaten Diagramme des Irisdatensatzes zusammen mit den jeweiligen Korrelationskoeffizienten dargestellt.
Abbildung 5-12: Bivariate Verteilungen mit Pearson-r des Irisdatensatzes
Starke Korrelation zwischen einem Feature und dem Target ist ein Indikator dafür, dass das Feature informativ oder relevant ist. Das bedeutet, dass es wertvolle Informationen besitzt, die ein Algorithmus zur Klassifizierung oder Regression verwenden kann.
Besteht eine starke lineare Korrelation oder Antikorrelation zwischen Feature und Target-Wert, besitzt das Feature eine hohe Klassifizierungsfähigkeit. In Abbildung 5-10 haben wir bereits gesehen, dass die Irisarten setosa und versicolor perfekt zu trennen sind. Dementsprechend erwarten wir auch einen hohen Korrelationswert zwischen dem Feature petal length und Target sowie petal width und Target, was wir mit folgendem Code überprüfen:
from sklearn.datasets import load_iris
import numpy as np
from scipy.stats import pearsonr # pearson package from scipy
iris = load_iris() # reload data
X = iris.data
y = iris.target
for fid in (0, 1, 2, 3): # loop over all features
idx = np.where( (y == 0) | (y == 1) )
x = X[idx]
x = x[:, fid]
print(iris.feature_names[fid], pearsonr(x, y[idx])[0])
Der Output sieht dann so aus:
sepal length (cm) 0.728290148746
sepal width (cm) -0.684019406159
petal length (cm) 0.969955270125
petal width (cm) 0.960157545411
Ein Pearson-Korrelationskoeffizient nahe null heißt nicht zwangsläufig, dass gar keine Beziehung zwischen den beiden Variablen besteht, sondern nur keine lineare, etwa bei einer quadratischen Beziehung:
x = np.random.uniform(-1, 1, 1000)
print(pearsonr(x, x**2)[0])
> 0.0187853623787
Dies ist auch einer der größten Nachteile des Pearson-Korrelationskoeffizienten. In Abbildung 5-11 oben siehst du ein paar Beispiele für verschiedene bivariate Verteilungen und die entsprechenden Pearson-r-Werte. In der untersten Reihe sind einige typische Verteilungen aufgeführt, die einen r-Wert von null besitzen, obwohl eine deutliche Beziehung zwischen x- und z-Wert besteht.
In dieser Beziehung etwas robuster ist die sogenannte Distance Correlation, die du hier nachschlagen kannst: https://en.wikipedia.org/wiki/Distance_correlation.
Abbildung 5-13 zeigt, dass der Distance-Korrelationskoeffizient bei den meisten Problemfällen einen von null verschiedenen Wert besitzt.
Abbildung 5-13: Beispiele für Distance-Korrelationskoeffizienten (Abbildung aus https://en.wikipedia.org/wiki/Distance_correlation)
Bei der Principal-Component-Analyse (PCA) benutzt man die Varianz der Daten als Kriterium zur Sortierung der Features. Die Idee dabei ist, die Koordinatenachse im Feature-Raum mit der größten Varianz zu finden. Die weiteren Achsen werden nach abnehmender Varianz sortiert mit der Einschränkung, dass alle Achsen orthogonal zueinander sein müssen. Auf diese Weise erhält man ein ganz neues Koordinatensystem und damit einen ganz neuen Feature-Raum.
Abbildung 5-14 zeigt eine zweidimensionale Verteilung zweier zufälliger Features x1 und x2. Eingezeichnet sind auch die berechneten PCA-Komponenten, skaliert mit der jeweiligen Varianz.
Transformiert man die Daten in den neuen Raum, der von den beiden PCA-Komponenten aufgespannt wird, erhält man das in Abbildung 5-15 Dargestellte.
Abbildung 5-14: PCA-Komponenten von zwei Features
Abbildung 5-15: Mit PCA transformierte Daten
PCA wird häufig bei der Datenexploration und -visualisierung benutzt, weil man hochdimensionale Daten in zwei- oder dreidimensionale Daten mit möglichst geringem Informationsverlust transformieren kann. Dadurch kann man z.B. visuell untersuchen, ob sich die Daten in bestimmten Bereichen clustern.
Beispielsweise können wir die vier Feature-Dimensionen des Irisdatensatzes mithilfe von PCA auf zwei Dimensionen reduzieren, visualisiert in Abbildung 5-16.
Abbildung 5-16: Die ersten beiden PCA-Komponenten der Irisdaten
Es gibt verschiedene Strategien zur Reduzierung der Input-Features. Bei den meisten Methoden wird jeweils eine Rangliste der Features aufgestellt, die für das gegebene Problem den vermeintlich höchsten Informationsgehalt besitzen. Drei der gängigsten Techniken stellen wir dir in diesem Abschnitt vor.
Im vorigen Abschnitt haben wir gesehen, dass die Korrelation bzw. die Varianz zwischen einem Feature und dem Target als Kriterium benutzt wird, um ein informatives Feature von einem weniger informativen Feature zu unterscheiden.
Die Varianz zwischen Features und Target können wir ausnutzen, um Features zu selektieren. Beim sogenannten F-Test wird die Varianz eines Features innerhalb der Klassen und zwischen allen Klassen berechnet, und der sich daraus ergebende Wert nennt sich F-Score:

Ein hoher F-Score ist ein Indikator für eine hohe Klassifizierungskraft eines Features.
Als Referenz berechnen wir zunächst den Score des linearen SVM-Klassifikators für die vier ursprünglichen Features:
from sklearn.datasets import load_iris
from sklearn.svm import SVC
import numpy as np
iris = load_iris()
X = iris.data
y = iris.target
# reference score
svc = SVC(kernel='linear', C=1)
clf = svc.fit(X, y)
print(clf.score(X, y))
> 0.993333333333
Der erhaltene Referenz-Score ist 0.99333. Danach generieren wir ein neues nicht-informatives Feature mithilfe des Zufallsgenerators in NumPy:
# Add random noise as non informative data
rns = np.random.RandomState(12)
noise = rns.uniform(0, 6, size=(len(X), 1))
X = np.hstack([X, noise])
clf = svc.fit(X, y)
print(clf.score(X, y))
> 0.993333333333
Wie wir sehen, hat sich der Score mit dem zusätzlichen Feature nicht geändert. Das neue Feature enthält eben keine relevante Information, es ist deshalb überflüssig.
Wie können wir nun generell solche Feature-Kandidaten detektieren und aus unserer Feature-Liste ausschließen? Die Sklearn-Bibliothek f_classif besitzt die Funktion, eine Rangliste der Features nach dem F-Score zu berechnen. Mit SelectKBest können dann die vier besten Features mit der Option k=4 ausgefiltert werden:
from sklearn.feature_selection import SelectKBest
from sklearn.feature_selection import f_classif
selector = SelectKBest(f_classif, k=4)
X_sel = selector.fit_transform(X, y)
Die F-Scores aller fünf Features kannst du mit print(selector.scores_) ausdrucken:
print(selector.scores_)
>
[ 1.19264502e+02 4.73644614e+01 1.17903433e+03 9.59324406e+02 9.48960689e-01]
Wie erwartet, hat das fünfte Feature den geringsten Score. Wir überprüfen das Ergebnis, indem wir den linearen SVM-Klassifikator wieder auf die selektierten Features anwenden (X_sel):
svc = SVC(kernel='linear', C=1)
clf = svc.fit(X_sel, y)
print(clf.score(X_sel, y))
Der Klassifizierungs-Score ist wiederum 0.99333. Das ergibt Sinn, da wir ja nun wieder bei dem ursprünglichen Feature-Satz sind.
Eine Auswahl der Features auf Basis der Varianz ist häufig nicht optimal, da nicht-lineare Beziehungen zwischen den Features und dem Target nicht berücksichtigt werden.
Die Anwendung von ML-Modellen zum Sortieren der Features hat diese Einschränkung nicht. Man kann z.B. ein Feature-Ranking mit einem RandomForest-Klassifikator aufstellen, den wir schon im vorherigen Kapitel kennengelernt haben. Bei DecisionTree-basierten Modellen wird bei jedem Branch-Split berechnet, wie viel ein Feature zur Minimierung der Impurity eines Zweigs beiträgt. Im Fall eines RandomForest-Klassifikator werden die Beiträge der verschiedenen Features gemittelt und im Attribut feature_importance gespeichert:
from sklearn.feature_selection import SelectFromModel
from sklearn.ensemble import RandomForestClassifier
clf = RandomForestClassifier()
clf.fit(X, y)
print(clf.feature_importances_)
Die Importances der fünf Features sind bei unserem Durchlauf folgende:
[ 0.1632587 0.02530171 0.26785365 0.52779362 0.01579231]
Je höher der Wert, desto größer ist die Wichtigkeit des Features. Diese Ergebnisse sind nicht deterministisch, wenn du das Beispiel ausprobierst, kannst du durchaus andere, aber ähnliche Ergebnisse bekommen.
Mit SelectFromModel können dann mithilfe der Option threshold (Schwellenwert) die Features nach feature_importance gefiltert werden:
selector = SelectFromModel(clf, threshold=0.02)
X_sel = selector.fit_transform(X, y)
print(selector.get_support())
Der Filter-Output ist ein boolesches Array, bei dem jeder Wert angibt, ob sich das Feature oberhalb (True) oder unterhalb (False) der threshold-Option befindet:
[ True True True True False]
Bei uns ist nur das letzte, künstlich hinzugefügte Feature unterhalb des Schwellenwerts.
Wir überprüfen das Ergebnis wieder, indem wir den linearen SVM-Klassifikator auf die selektierten Features anwenden (X_sel):
svc = SVC(kernel='linear')
clf = svc.fit(X_sel, y)
print(clf.score(X_sel, y))
Der Score lautet wie erwartet:
0.993333333333
Ein Beispiel für einen generelleren Einsatz von ML-Modellen ist die iterative Eliminierung von Features. Dabei wird nach jeder Iteration dasjenige Feature eliminiert, das am wenigsten zur Klassifizierung oder Regression beiträgt. Wir sehen uns hier als Beispiel einen Random Forest-Klassifikator an, bei dem die besten vier Features selektiert werden.
Zunächst trainieren wir RandomForestClassifier mit den Daten:
from sklearn.ensemble import RandomForestClassifier
clf = RandomForestClassifier()
clf.fit(X, y)
Dann wird eine Instanz der Recursive-Feature-Elimination-Klasse erzeugt. Als Klassifizierungsmethode geben wir unseren Random ForestClassifier an. Die Anzahl der zu selektierenden Features wird auf 4 gesetzt:
from sklearn.feature_selection import RFE
selector = RFE(clf, 4)
Jetzt wird der Selektor trainiert:
selector = selector.fit(X, y)
Und schließlich wird das daraus resultierende Feature-Filter-Array ausgedruckt:
print(selector.get_support())
[ True True True True False]
Dieses Array kann wieder genau wie im vorigen Beispiel benutzt werden, um die Features zu filtern.